유전체 압축 및 저장 표준 동향
Trends of Standardization for Genome Compression and Storage
- 저자
- 정순흥, 박수준, 김휘용, 최진수 / 실감AV연구그룹
- 권호
- 32권 1호 (통권 163)
- 논문구분
- 일반 논문
- 페이지
- 116-122
- 발행일자
- 2017.02.01
- DOI
- 10.22648/ETRI.2017.J.320112
 본 저작물은 공공누리 제4유형: 출처표시 + 상업적이용금지 + 변경금지 조건에 따라 이용할 수 있습니다.
본 저작물은 공공누리 제4유형: 출처표시 + 상업적이용금지 + 변경금지 조건에 따라 이용할 수 있습니다.- 초록
- 유전체 분석을 위한 시퀀싱 기술의 발전으로 유전체 데이터량이 폭발적으로 증가하고 있다. 저장 및 관리 비용 절감을 위해 유전체 데이터 압축 기술이 연구되고 있지만, 국제 표준의 부재로 다양한 포맷들이 사용되고 있다. 최근, MPEG에서 유전체 데이터의 압축 및 저장 표준에 대한 필요성을 받아들여 표준화 작업이 진행 중이다. 본고에서는 유전체 분석의 기본이 되는 염기서열의 분석 과정을 소개하고, 유전체 데이터 압축 및 저장 기술의 표준화 동향에 대해서 살펴보고자 한다.
Share
Ⅰ. 서론
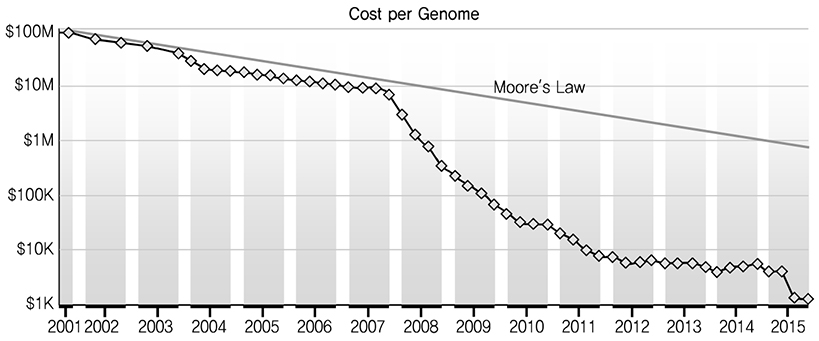
인간의 유전체 지도를 해독하기 위해 13년간 진행된 인간 지놈 프로젝트(Human Genome Project)가 2003년 종료된 이후, 유전체 분석을 위해 필요한 비용은 기하 급수적으로 감소하였다. 미국 NIH(National Institutes of Health)의 발표 자료는 2001년 1억달러에 달하던 인간의 유전체 분석 비용이 2015년 이후 1000달러 수준으로 줄어들고 있음을 보여준다[(그림 1) 참조]. 이러한 비용의 감소는 유전체 관련 산업의 발전을 이끌고 있으며, 분석된 대용량의 유전체 데이터는 빅데이터 기술과 맞물려 유전체 분석 시장의 발전을 가속화하고 있다.
(그림 1)
무선인터넷 실태조사

<출처>: NIH, “The Cost of Sequencing a Human Genome” 2016. 6. (https://www.genome.gov/sequencingcosts/)
Google과 Amazon은 보유하고 있는 클라우드 시스템을 활용하여 대용량의 유전체 데이터를 저장, 처리, 분석할 수 있는 클라우드 서비스를 제공하고 있으며, 100만명 이상이 23andme를 통해서 유전체 분석 서비스를 이용하였다[1][2]. 2014년 미국의 Broad Institute와 중국의 BGI(Beijing Genomics Institute) 등 대규모 유전체 연구소에서 하루에 생성되는 데이터의 양은 수 백 테라바이트 급에 달했으며, 미국 국립암센터(National Cancer Institute)는 2.6 페타바이트(petabyte)의 암 유전체 정보를 클라우드로 복사하는데 1,900만 달러를 지출할 것을 발표하였다[3].
이와 같이 급격하게 증가하는 대용량 유전체 데이터의 저장 및 관리 비용 감소를 위한 압축 기술의 필요성이 수년 전부터 꾸준히 제기되어 왔다[4]. 최근에는 국제 표준화 기구인 ISO/IEC 산하 MPEG(Moving Picture Experts Group)에서 유전체 데이터 압축 및 저장 기술에 대한 표준의 필요성을 받아들여 표준화가 진행 중이다. 본고에서는 유전체 분석의 기본이 되는 염기서열의 분석 과정을 소개하고, 유전체 데이터 압축 및 저장 기술의 표준화 동향에 대해서 살펴보고자 한다.
Ⅱ. 유전체 개요
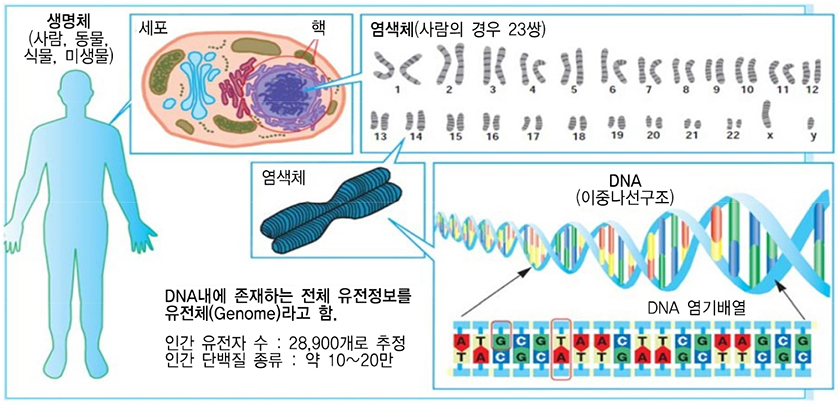
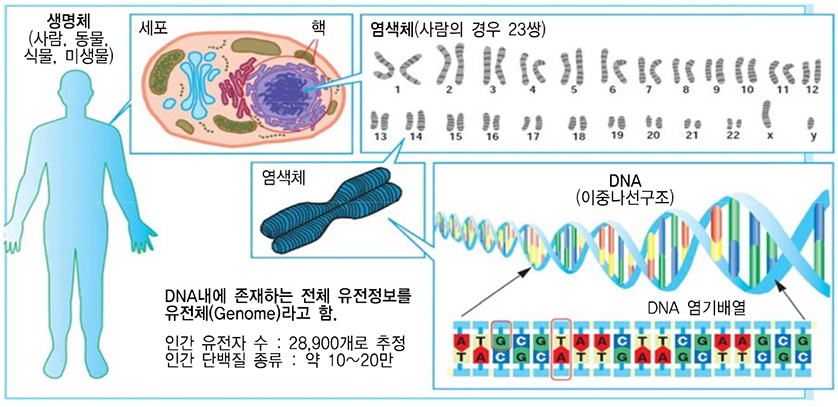
유전체(genome)란 유전자(gene)와 염색체(chromosome)의 합성어로 개체를 만드는 데 필요한 모든 유전자와 유전자 바깥 부분을 포함하는 DNA(Deoxyribonucleic Acid) 염기서열을 통틀어서 말한다[(그림 2) 참조]. DNA는 세포 내의 염색체 안에 이중나선구조의 형태로 존재하며, A(Adenine), T(Thiamine), G(Guanine), C(Cytosine)의 염기들이 A-T, G-C의 쌍들로 이루어져 있다. 인간은 23쌍의 염색체를 가지며, DNA 내에는 약 32억쌍의 염기서열이 존재하는 것으로 알려져 있다.
인간의 유전체는 99% 이상이 일치하며, 1% 미만의 차이로 서로 다른 모습을 갖게 된다. 이러한 염기서열의 정보는 정밀의료, 신약 개발, 농업 및 동물 연구에 활용되고 있다. 2016년 MIT 10대 혁신기술에 3개의 유전체 관련 기술(면역공학, 농작물 유전자 에디팅, DNA 앱스토어)이 포함되는 것으로 미루어 보면, 미래의 유전체 산업에 대한 전망이 상당히 밝다고 볼 수 있다[5].
Ⅲ. 염기서열 분석 기술 및 장비 현황
유전체 분석을 위해서는 염기서열 분석 과정이 필요하다. 염기서열 분석 기술로는 생어 시퀀싱 기술(Sanger sequencing)과 차세대 염기서열 분석 기술(NGS: Next-generation DNA sequencing)이 많이 사용된다[6]. 생어 시퀀싱 기술은 분석의 정확도 면에서 성능이 우수하지만, 비용과 속도의 문제로 인해 차세대 염기서열 분석 기술이 대중적으로 사용되고 있다.
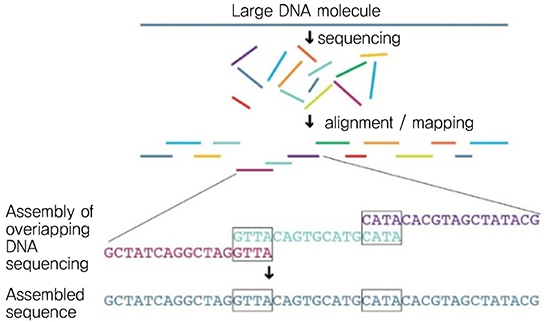
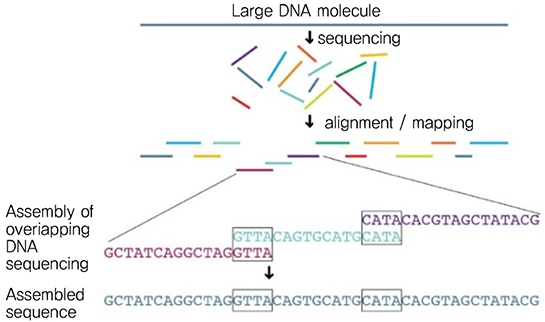
유전체 염기서열 분석은 DNA를 무작위 조각인 read로 나누어 염기서열 정보를 분석하고, 이를 조합하여 전체 유전체의 염기서열 정보를 생성하는 과정으로 이루어진다. (그림 3)은 read 단위의 유전체 시퀀싱 과정을 보여준다[7]. Read는 100~20000개 이상의 base(염기: A, T, G, C)로 구성될 수 있으나, 길어질수록 분석의 정확도가 떨어지는 경향을 갖는다.
(그림 4)는 시퀀싱 이후 read를 이어붙여 전체 염기서열 정보를 만들어내는 과정의 예를 보여준다. (그림 4)와 같이 참조 염기서열의 정보가 존재하지 않는 경우, 시퀀싱된 read간 중복되는 정보 등을 활용하여 전체 염기서열 정보를 생성하게 된다. 따라서 많은 양의 동일한 유전체가 read로 나누어져 시퀀싱 될수록, 조합된 전체 염기서열 정보의 정확도가 높아지게 된다. 일반적으로 임상용을 위해서는 200개 이상의 동일한 유전체 시퀀싱이 필요하다. 참조 염기서열의 정보가 존재하는 경우에는 시퀀싱된 read의 염기서열 정보와 매핑되는 부분을 활용하여 전체 염기서열 정보를 생성한다.
인간의 염기는 4가지로 이루어져 있으므로 하나의 염기는 2bits로 표현이 가능하다. 32억쌍의 염기 정보를 저장하기 위해서는 32억×2bits=64억bits, 즉 800MB의 저장 공간이면 충분하다. 하지만 차세대 염기서열 분석기로 시퀀싱된 read는 시퀀싱 데이터 조합을 위해 염기정보 외 염기당 정확도(quality score)와 부가정보를 포함할 수 있다. 그리고 정확도가 떨어지는 염기는 A, T, G, C외 다른 코드로 표기하기도 한다. 이 때문에 임상용으로 시퀀싱된 read 정보를 저장하기 위해서는 약 1.5TB의 저장 공간이 필요하게 된다[7].
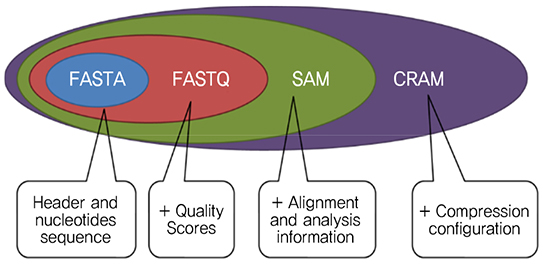
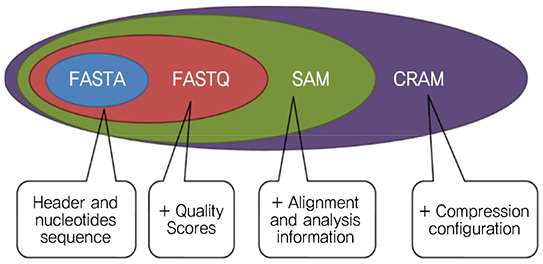
한편, 시퀀싱된 정보를 저장하기 위해서 (그림 5)와 같이 FASTA, FASTQ, SAM, CRAM의 4가지 파일 포맷이 많이 사용된다[8]. FASTA는 헤더와 read 단위의 분석된 염기서열 정보를 payload로 가진다. FASTQ는 FASTA 정보에 정확도(quality score) 정보가 추가된 파일 포맷이다. SAM은 FASTQ 정보에 read 정렬 및 분석 정보가 추가된 것이며, CRAM은 SAM을 압축한 파일 포맷이다.
차세대 염기서열 분석 장비는 제조사 및 모델에 따라 분석에 필요한 read 길이가 다를 수 있고, 분석 속도 및 정확도 등에서 차이가 있으며, 수만에서 수십만 달러의 가격대를 형성하고 있다[9]. 이 중에서, Illumina사의 플랫폼이 세계적으로 가장 많이 사용되어 GenBank에 등록된 염기서열의 90% 가량을 차지하고 있다[3]. 2014년 5월 국가연구시설장비진흥센터의 자료에 의하면 한국에서도 high performance급 장비는 Illumina 플랫폼이 53대로 전체의 70.7%를 차지하였으며, benchtop급 장비는 LT(현재는 Thermo Fisher)사 장비가 23대로 전체의 45.1%, 그 뒤로 Illumina 장비가 18대로 35.3%를 차지하였다[3].
Ⅳ. 유전체 데이터 압축 및 저장 기술 표준화 동향
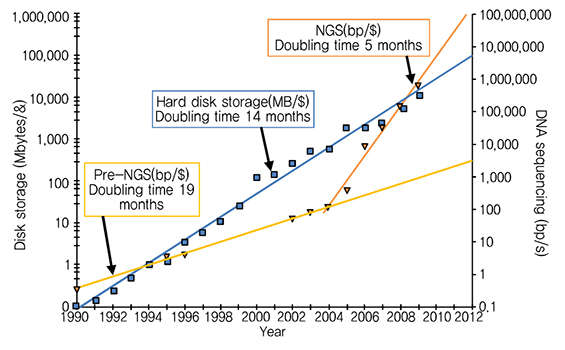
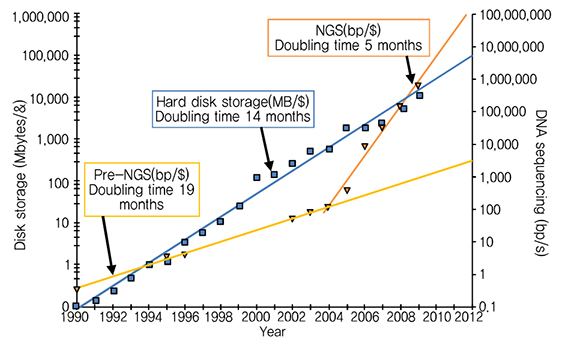
서론에서 언급한 바와 같이 유전체 시퀀싱에 필요한 비용 절감이 급격하게 이루어지면서, (그림 6)과 같이 2004년 이후 NGS 장비의 달러당 유전체 분석량 증가 추세는 달러당 디스크 용량 증가 추세를 추월하였다[10]. 이러한 추세를 누그러뜨리기 위한 방법으로 유전체 데이터 압축 기술에 대한 연구가 꾸준히 진행되고 있으나, 관련된 국제 표준은 아직 마련되지 못한 상황이다.
2014년 3월 스페인의 발렌시아에서 진행된 108차 MPEG 회의에서 유전체 데이터의 압축, 저장, 그리고 스트리밍에 대한 필요성이 최초로 제기되었다[11]. 해당 기고서는 스위스의 두 학교와 한 연구소(EPFL: Ecole Polytechnique Federale de Lausanne), HES-SO(Haute école spécialisée de Suisse occidentale), SIB(Swiss Institute of Bioinformatics)에 속한 비디오 압축 전문가와 바이오 인포매틱스 전문가에 의해 공동으로 작성되었다. EPFL과 HES-SO는 모두 SIB의 기관회원이다. 전통적으로 오디오와 비디오 데이터의 압축과 처리에 대한 표준화를 진행해온 MPEG에 유전체 데이터에 대한 표준 이슈를 제기한 것은 특이하게 생각할 수 있지만, 데이터의 종류를 확장하면 MPEG에서 다룰 만한 표준이라고 볼 수도 있다. MPEG에서 관련 표준의 필요성을 받아들였고, 이어지는 회의에서 산학연이 추가로 참여하여 2016년 10월까지 20여개 기관이 참여하고 있다.
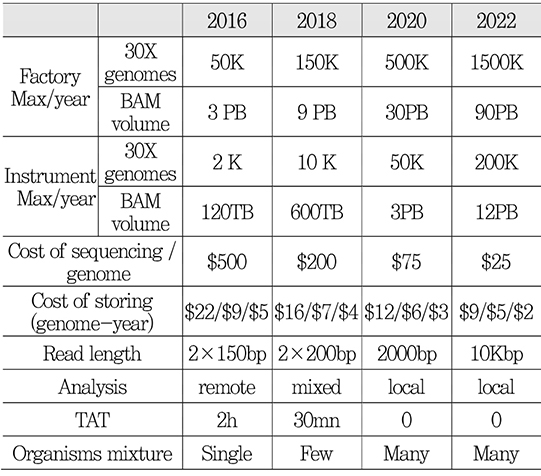
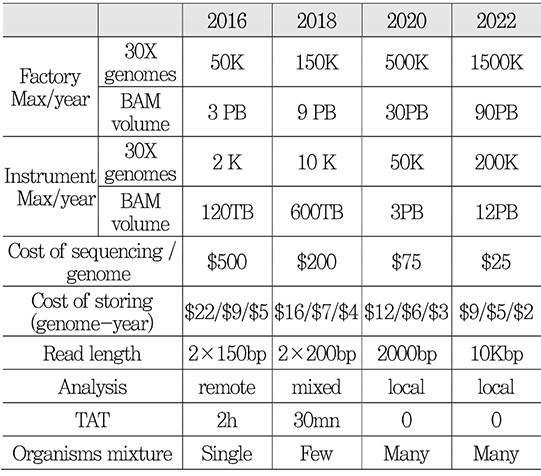
2016년 2월 미국 샌디에고에서 진행된 114차 MPEG 회의에서 유전체 압축 및 저장 표준의 CfE(Call for Evidence)에 대한 평가가 이루어졌다. 그 결과, 최신 기술보다 27%이상 압축율을 높일 수 있음을 확인하고 표준화를 진행하기로 결정하였다[12]. 이 회의에서 유전체 압축에 대한 세미나도 진행되었는데, 세계 1위의 NGS 장비 생산 업체인 Illumina와 생명공학(Biotech-nology) 분야 표준화를 맡고 있는 ISO/TC 276의 WG5 Co-convenor Yong Zhang 등이 유전체 데이터의 압축 필요성과 고려사항에 대해 발표하였다. 특히, Illumina는 <표 1>과 같이 factory 및 instrument 규모의 유전체 분석 데이터량 예상치를 제시하였는데, 2022년에는 factory에서 연간 150만명에 대한 유전체를 분석하여 90 peta bytes의 BAM 파일을 생성할 수 있을 것으로 예상하였다[13]. 또한, 시퀀싱 비용이 2022년에 20배로 감소하는 데 비해 저장 비용은 최대 2.5배 감소하여 저장에 필요한 비용의 비율이 높아질 것으로 전망하였다[13].
2016년 6월에 스위스 제네바에서 진행된 115차 MPEG 회의를 통하여 ISO/TC 276과 공동으로 유전체 압축 및 저장 표준에 대한 JCfP(Joint Call for Proposal)를 발행하였다[14]. 요구사항은 read 단위 압축에 대한 요구사항 9개, 시퀀싱 데이터 조합 과정에서 생성되는 맵핑 또는 정렬된 read에 대한 압축 요구사항 9개, 마지막으로 전송에 대한 요구사항 5개, 총 23개로 구체화 되었다[15]. 유전체의 염기서열 자체에 대해서는 무손실 압축, 정확도(quality score)와 일부 부가 데이터에 대해서는 손실 압축을 허용하고 있으며, 응용을 고려한 임의접근 등의 요구사항이 포함되었다. 요구사항에 대한 평가 방법과 사용할 데이터에 대한 정보는 문서로 제공되고 있다[16][17].
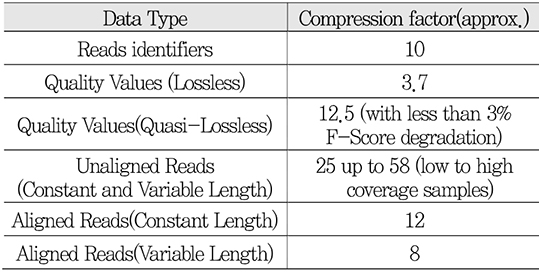
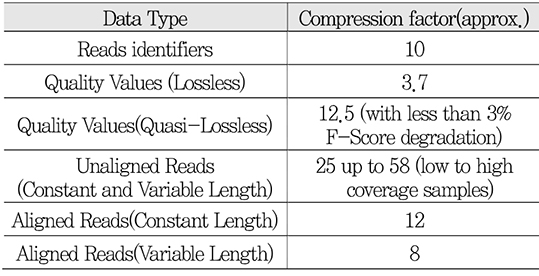
2016년 10월 중국 청두에서 진행된 116차 MPEG 회의에서는 CfP에 대응한 15개의 기고 문서에 대한 평가와 이에 따른 CE(Core Experiment) 수립이 진행되었다. 9건의 단독 기고와 6건의 공동 기고가 있었으며, 3개의 회사, 8개의 대학, 6개의 유전체 연구기관이 참여하였다. 평가결과, 제안 기술들을 조합하여 JCfP 요구사항의 모든 항목을 만족시킬 수 있다고 판단하였으나, 전체 테스트 데이터에 대한 결과를 제출하지 못한 경우가 많아 CE를 통해 제안 기술들을 모두 재검토하고 재실험하여 항목별로 가장 좋은 성능을 가지는 기술을 선별하기로 하였다. 또한, 회의 중 테스트 콘텐츠의 문제점과 보완이 필요한 부분들이 나타남에 따라 CE 진행 시 사용할 데이터를 재정의하고, 추가 고려사항을 반영하기로 하였다. 제안된 기술들을 기반으로 평가된 데이터 종류에 따른 압축 성능은 <표 2>와 같다[18].
전송 관련 5가지 요구사항을 모두 만족시키는 기술은 GenomSys사에서 제안하는 HEGIC(High-Efficiency Genomic Information Coding) 기술과 8개 기관에서 공동 기고한 GENIFF(GENomic Information File Format) 기술 2가지였다[19][20].
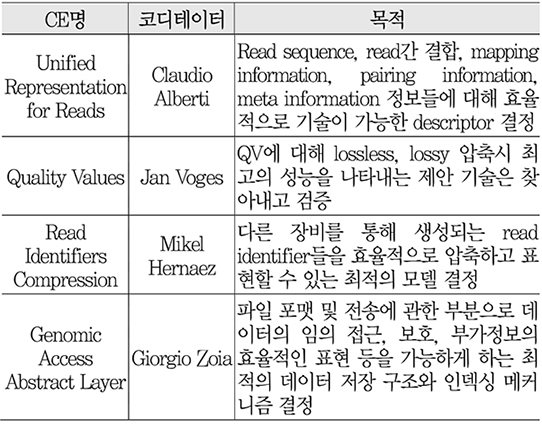
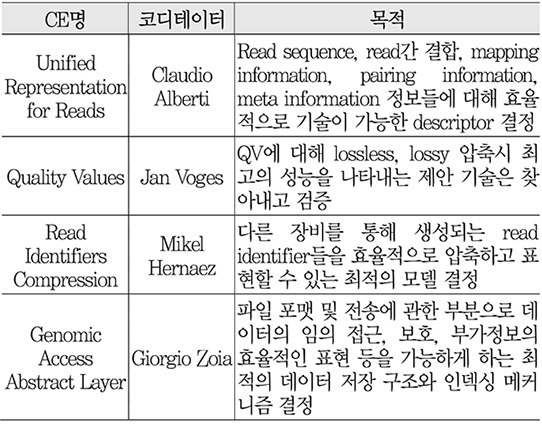
제안된 기술들은 이번 회의에서 수립된 <표 3>의 CE를 통해 검증 과정을 거쳐 요구사항에 따라 가장 우수한 성능을 가지는 기술이 선별될 예정이다[21].
이후 2017년 1월에 draft 초안과 test model 초안 작성, 2017년 10월에 CD(Committee Draft), 2018년 7월에 DIS(Draft International Standard), 2019년 1월에 FDIS(Final Draft International Standard)를 발행하는 것으로 일정이 계획되어 있다[18].
Ⅴ. 결론
본고에서는 유전체 압축에 대한 필요성과 현재까지 진행된 유전체 압축 및 저장에 대한 표준화 동향에 대해서 설명하였다. MPEG과 ISO/TC 276이 공동으로 최초의 유전체 압축 및 저장에 대한 표준화 작업을 진행 중이며, 2016년 10월 진행된 JCfP 대응에 대한 평가 결과 제안된 기술들의 조합을 통해서 전체 요구사항을 만족시킬 수 있을 것으로 예상한다.
한편, 현재 NGS 기술은 2세대 기술로 3세대 시퀀싱 기술 개발이 진행 중이며, 3세대 시퀀싱 기술로 얻어지는 데이터의 형태는 달라질 수 있으므로 관련 표준이 이에 맞게 적절하게 보완될 필요가 있을 것으로 보인다. 시퀀싱 기술이 발달하여 한번에 염기서열 정보를 정확하게 해석할 수 있게 된다면, 32억쌍에 해당하는 800MB의 정보만 잘 압축하면 되므로 압축에 대한 필요성이 낮아지거나 없어질 수도 있다. 하지만 이러한 기술이 출현하기까지는 시간이 필요할 것으로 예상되며, 출현하더라도 기존에 분석된 데이터의 활용 측면에서 해당 표준 기술은 여전히 유효하게 사용될 수 있을 것이다.
유전체 압축 및 저장 표준에 대한 주요 수요는 Google, Amazon 등 유전체 데이터를 대용량으로 보유하고 있는 업체, 유전체 센터 및 연구 기관, 병원 등에서 발생할 것으로 예상되며, 일반인에게까지 사용되는 동영상 표준에 비해서 규모는 제한적일 것으로 보인다. 하지만, 유전체 데이터량의 증가율을 고려하면 효율적인 압축 및 저장 기술을 통해 관련 비용을 줄여줄 필요성은 분명해 보인다. 또한, 동물과 식물에 대한 유전체 분석과 네트워크를 통한 전송까지 고려하면 관련 기술에 대한 필요성은 더욱 커질 수 있을 것으로 예상된다.
용어해설
시퀀싱(Sequencing)염기서열 정보를 해석하는 기술
약어 정리
A
Adenine
BGI
Beijing Genomics Institute
C
Cytosine
CD
Committee Draft
CE
Core Experiment
CfE
Call for Evidence
DIS
Draft International Standard
DNA
Deoxyribonucleic Acid
EPFL
Ecole Polytechnique Federale de Lausanne
FDIS
Final Draft International Standard
G
Guanine
GENIFF
GENomic Information File Format
HEGIC
High-Efficiency Genomic Information Coding
HES-SO
Haute ecole specialisee de Suisse occidentale
JCfP
Joint Call for Proposal
MPEG
Moving Picture Experts Group
NGS
Next-generation DNA sequencing
NIH
National Institutes of Health
SIB
Swiss Institute of Bioinformatics
T
Thiamine
Scott D. Kahn, “On the Future of Genomic Data,” Science, vol. 331, Feb. 11th, 2011, pp. 728-729.
J. Shendure and H. Ji, “Next-Generation DNA Sequencing,” Nature biotechnology, vol. 26, No. 10, Oct. 2008.
C. Alberti and M. Mattavelli, “Genome Compression 101 - Tutorial on Genome Compression and Storage,” ISO/IEC JTC 1/SC 29/WG 11 N15527, June, 2015.
“Database for Evaluation of Genomic Information Compression and Storage,” ISO/IEC JTC 1/SC 29/WG 11 N16145, Feb. 2016.
C. Alberti et al., “Proposal for Opening an Exploration Activity within SC29WG11 for the Definition of a Standard Technology for the Compression, Storage and Streaming of Genome Data,” ISO/IEC JTC 1/SC 29/WG 11 M33114, Mar.-April, 2014.
“Results of the Evaluation of the CfE on Genomic Information Compression and Storage,” ISO/IEC JTC 1/SC 29/WG 11 N16147, Feb. 2016.
“Joint Call for Proposals for Genomic Information Compression and Storage,” ISO/IEC JTC 1/SC 29/WG 11 N16320, ISO/TC 276/WG 5 N99, June, 2016.
“Requirements on Genomic Information Compression and Storage,” ISO/IEC JTC 1/SC 29/WG 11 N16323, ISO/TC 276/WG 5 N97, June, 2016.
“Evaluation Procedure for the Joint Call for Proposals on Genomic Information Compression and Storage,” ISO/IEC JTC 1/SC 29/WG 11 N16321, ISO/TC 276/WG 5 N98, June, 2016.
“Database for Evaluation of Genomic Information Compression and Storage,” ISO/IEC JTC 1/SC 29/WG 11 N16322, ISO/TC 276/WG 5 N96, June, 2016.
“Summary of the Current Status and Workplan of the Joint TC276/WG5 JTC1/SC29/WG11 Standardization Activities on Genomic Information Representation,” ISO/IEC JTC 1/SC 29/WG 11 N16529, ISO/TC 276/WG 5 N122, Oct. 2016.
G. Zoia and D. Renzi, “Coding and Transport Framework for Genomic Information,” ISO/IEC JTC 1/SC 29/WG 11 M38961, Oct. 2016.
J. Delgado et al., “GENIFF (GENomic Information File Format), a proposal for a Secure Genomic Information Transport Layer (GITL) based on the ISO Base Media File Format,” ISO/IEC JTC 1/SC 29/WG 11 M39175, Oct. 2016.
- Sign Up
- 전자통신동향분석 이메일 전자저널 구독을 원하시는 경우 정확한 이메일 주소를 입력하시기 바랍니다.